COVID-19 SEIR 모델
개요
SEIR 모델을 COVID-19의 특성을 반영한 공간에서 구현
SEIR 모델은 기존 SIR 모델을 개선하여 제작된 수학적 역학(epidemiology) 모델
S : susceptible (감염취약군)
E : exposed (노출군)
I : infectious (감염군)
R : recovered (회복군)
그렇기에 각 군은 시간에 종속인 함수로 볼 수 있으므로 S(t), E(t), I(t), R(t)로 표현할 수 있고 다른 변수들을 소개하자면 다음과 같다
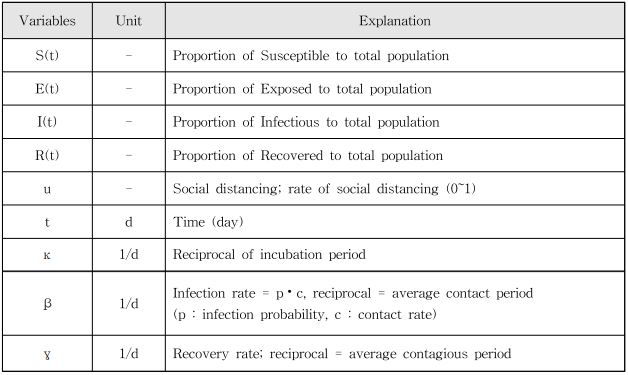
또한 각 군에 대해서는 다음의 관계가 성립한다
(ARC에 쓴건데 내 최악의 영어 실력을 감안해야함)

위 글에서 볼 수 있 듯이 4개의 군들의 관계는 상미분 방정식(ODE) 의 비선형시스템으로 얽혀있고
이것을 적분하여 푼다면 시간에 따른 각 군들의 변화의 추이를 볼 수 있다
그러나 그것을 어떻게 푸는지 모르는 1인 강준서,
scipy 모듈의 integrate.odeint 함수를 이용한다
밑과 같은 함수를 설정하여 각 군끼리의 관계를 설정항 다음 odeint함수에 집어넣는다
parameter로는 covid 함수, x(S,E,I,R의 정보가 리스트에 담겨있는 것), t(변수) 가 사용되었다
아래는 그냥 covid 함수 어떻게 생겼는지에 대한 것
def covid(x,t):
s,e,i,r = x
dx = np.zeros(4)
dx[0] = -(1-u)*beta*s*i
dx[1] = (1-u)*beta*s*i - alpha*e
dx[2] = alpha*e - gamma*i
dx[3] = gamma*i
return dx
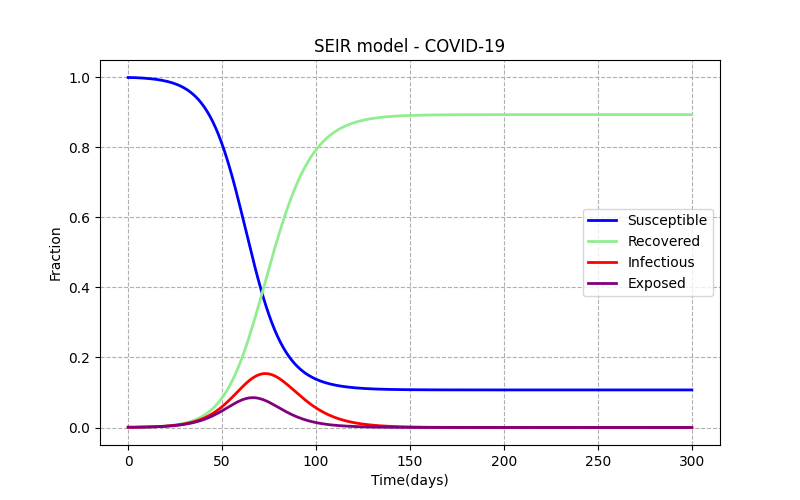
이렇게 해서 프로그램으로 적분 때리면 결과 항목의 그래프가 도출된다
초기 설정
R0 = 2.5
incubation period = 4
infectious period = 8
(COVID-19와 동일한 값으로 설정)
결과
소스코드
import numpy as np
from scipy.integrate import odeint
import matplotlib.pyplot as plt
u = 0.0 # social distancing
t_incubation = 4 # 평균 지연시간(일), 원래 잠복기 자체는 6일인데 증상 발현 이틀전부터 감염 가능성 확인, 4로 조정
t_infectious = 8
R0 = 2.5
N = 800 # population 시뮬레이션의 인구와 동일하게 지정
# initial number of infectious and recovered individuals
e0 = 1/N
i0 = 0.00
r0 = 0.00
s0 = 1 - e0 - i0 - r0
x0 = [s0,e0,i0,r0]
alpha = 1/t_incubation
gamma = 1/t_infectious
beta = R0*gamma
def covid(x,t):
s,e,i,r = x
dx = np.zeros(4)
dx[0] = -(1-u)*beta*s*i
dx[1] = (1-u)*beta*s*i - alpha*e
dx[2] = alpha*e - gamma*i
dx[3] = gamma*i
return dx
t = np.linspace(0,300,201)
x = odeint(covid, x0, t)
s = x[:,0]; e = x[:,1]; i = x[:,2]; r = x[:,3]
# 그래프
plt.figure(figsize = (8,5))
plt.title('social distancing = {}%'.format(u*100))
plt.plot(t,s,color = 'blue', lw = 2, label = 'Susceptible')
plt.plot(t,r,color = 'lightgreen', lw = 2, label = 'Recovered')
plt.plot(t,i, color = 'red', lw = 2, label = 'Infectious')
plt.plot(t,e, color = 'purple', lw = 2, label = 'Exposed')
plt.xlabel('Time(days)')
plt.ylabel('Fraction')
plt.grid(linestyle = '--')
plt.legend()
plt.show()